Configuración preliminar
conda config --add channels defaults
conda config --add channels bioconda
conda config --add channels conda-forge
Descargue los datos e instale el ambiente
gdown --folder https://drive.google.com/drive/folders/16He3-s3Vc3WQZ6dLmRU12Dm_aTe0Irpg?usp=sharing
cd clase_arboles
conda env create -f env-bayes.yml # Creará un ambiente usando información del archivo descargado.
conda activate bayes-tree
Alinear la secuencia con mafft
mafft --auto --thread 8 secuencias2.fasta > secuencias.aln.fasta
ls -lh
Cortar con trimal
trimal -automated1 -terminalonly -in secuencias.aln.fasta -out secuencias.trim.fasta
Una opción para seleccionar el modelo
modeltest-ng --help
modeltest-ng -i secuencias.trim.fasta -d nt -p 8 -T mrbayes -o secuencias.modeltest
Note que no hay una opción de modelo para iqtree, pero se podría usar el mejor modelo encontrado en algún otro programa.
Usar iqtree para generar el árbol
De igual forma, iqtree permite una opción para seleccionar el modelo en automático
iqtree -s secuencias.trim.fasta -m MFP -bb 1000 -nt AUTO
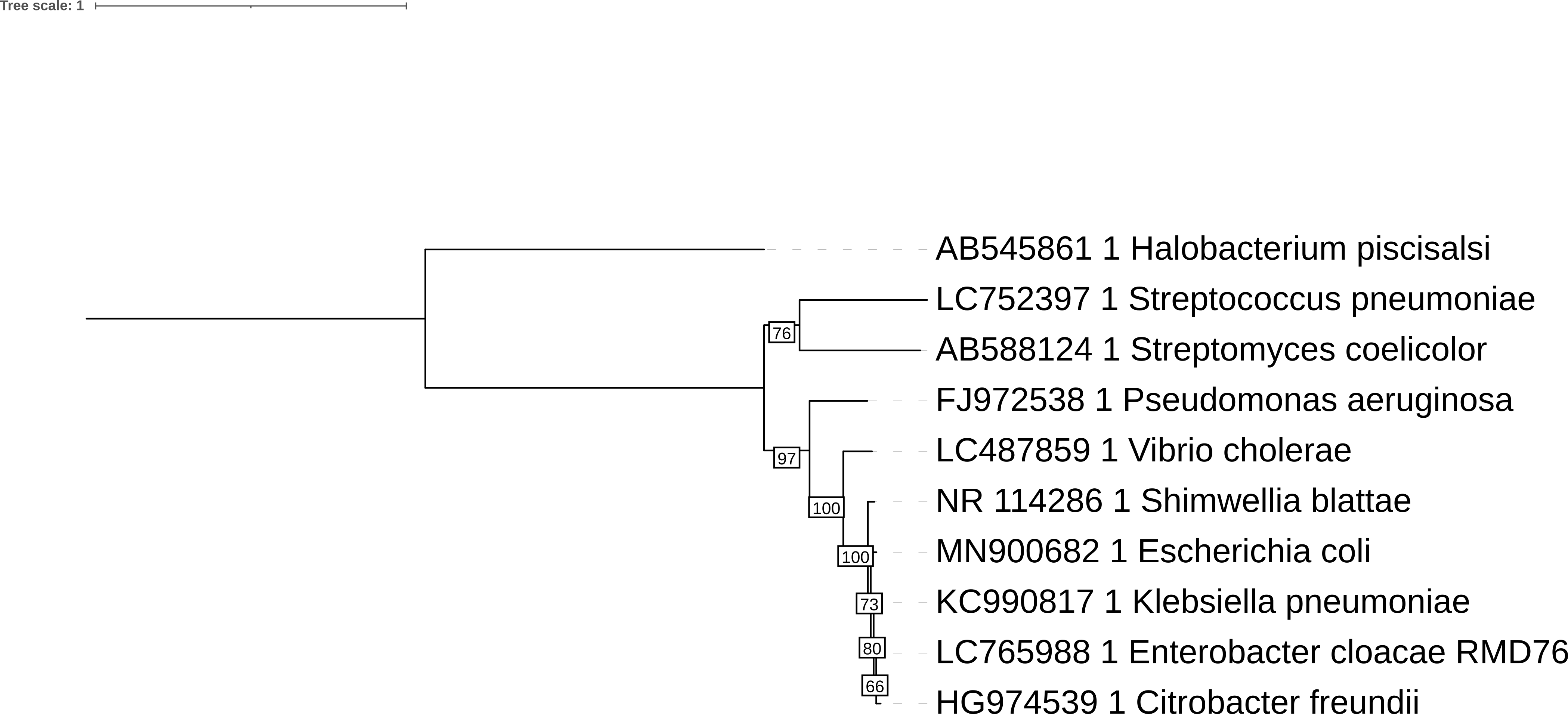
Visualice el árbol en iTOL